锁核苷酸PCR解析植物内生细菌多样性方法
Locked nucleic acid (LNA) oligonucleotide PCR clamping method for investigating the plant endophyte bacteria
于镇华1,Makoto Ikenaga2,Masao Sakai2,王光华1*
1中国科学院黑土区农业生态重点实验室,中国科学院东北地理与农业生态研究所,哈尔滨,黑龙江,150031
2日本鹿儿岛大学农学院,日本鹿儿岛,890-0065
*通讯作者邮箱: wanggh@iga.ac.cn
引用格式:于镇华, Makoto Ikenaga, Masao Sakai, 王光华. (2021). 锁核苷酸PCR解析植物内生细菌多样性方法. // 微生物组实验手册. Bio-101: e2003525. DOI: 10.21769/BioProtoc.2003525.
How to cite: Yunyun Gao, Kai Peng, Defeng Bai, et al. 2024. The Microbiome Protocols eBook initiative: Building a bridge to microbiome research. iMeta 3: e182. https://doi.org/10.1002/imt2.182
摘要
对植物内生细菌进行PCR扩增是内生细菌多样性研究的关键实验环节,对后续进一步解析植物内生细菌多样性和他们的生态功能具有重要意义。但是,由于植物线粒体和叶绿体SSU rRNA基因序列与细菌SSU rRNA基因序列存在高度的相似性,因此,采用传统的PCR扩增方法会带来大量源自植物细胞器扩增产物的污染,给后续的内生细菌群落结构分析带来干扰。为了克服这一难题,本方法采用锁核苷酸LNA(Locked Nucleic Acid)引物对植物细胞器SSU rRNA进行锚定,可以有效地遏制植物细胞器SSU rRNA基因的扩增,从而达到特异性扩增内生细菌的SSU rRNA基因的目的。该方法可与高通量测序方法结合起来获取更多植物内生细菌多样性信息。
关键词: 植物内生菌,锁核苷酸,LNA,PCR,SSU rRNA基因
材料与试剂
植物基因组DNA提取试剂盒(BioTeke, 货号DP3111)、High Pure PCR Product Purification Kit(Roche, Indianapolis IN, USA, Code No. 11732668001)、移液器、枪头、无菌水、PCR相关试剂(Takara RR030A Premix Ex Taq™ Hot Start Version, Code No. RR030A)、LNA扩增引物和细菌特异性扩增引物(华大公司合成)。
仪器设备
超纯水仪、LDZX-75KB立式压力蒸汽灭菌器、-20°C冰箱、BIO-RAD低温离心机、DYY-6C型电泳仪、超净工作台、PCR仪、Gel Doc凝胶成像系统、FM40制冰机。
实验原理:
本方法的设计原理如图1所示,主要是通过设计与常规细菌PCR引物有交叠序列的、针对于植物细胞器的特异LNA引物,由于LNA引物的Tm值较高,因此可在较高温度(70℃)时与植物细胞器基因退火,而常规细菌PCR引物不能完成此过程。之后,由于交叠部分的碱基位置被LNA引物锚定,在较低温度的Tm值时,如54℃时,常规PCR产物不能和植物细胞器基因退火,加之LNA引物的3’端引物被磷酸化,不能被用于PCR引物,PCR反应被终止,从而达到选择性的扩增细菌SSU rRNA基因,而抑制了植物细胞器基因扩增的目的。
图1 LNA-PCR工作原理简图
实验步骤
以提取样品DNA为模板,利用LNA-PCR引物(表1)进行第一轮PCR扩增,25 μL反应体系为:Premix(5 U·μL-1,ExTaq,Takara)12.5 μL、KU63f/KU1494r(20 pmol·μL-1)各1 μL、LNA-Mit63/LNA-Mit1492(20 pmol·μL-1)各3.75 μL、LNA-Pla63/LNA-Pla1492(20 pmol·μL-1)各1.25 μL、DNA模板0.5 μL、无菌水定容至25 μL。扩增条件为:94°C 3 min、94°C1 min、70°C1 min、54°C1 min、72°C2 min(30个循环)、72°C10 min。获得PCR产物后,将PCR产物进行琼脂糖电泳检测,电泳条件如下(供参考,可根据电压和电泳时间进行调整):琼脂糖浓度1-1.5%;电压120 V-150 V;marker(100 bp-1500 bp)。此步PCR反应的目的是抑制来自植物体本身细胞器的基因对PCR扩增的干扰,最大限度的提高来自植物内生菌基因的PCR扩增。研究者可以根据自身实验目的和需求,决定是否以第一步PCR扩增产物为模板进行第二步PCR反应。比如,想用DGGE技术来研究植物内生细菌多样性,那么,由于第一步PCR反应的产物片段较长,不适用于DGGE技术(片段长度最好小于500 bp),因此需要通过第二步PCR扩增缩短片段长度;如果想进行二代高通量测序,也需要进行第二步PCR扩增来缩小片段长度并使用带barcode的扩增引物。进行第二步PCR时首先要利用High Pure PCR Product Purification Kit(Roche, Indianapolis IN, USA, Code No. 11732668001)对上述PCR产物进行纯化,目的是对第一步PCR产物进行纯化稀释,去除第一步PCR产物中可能会对第二步PCR反应产生干扰的杂质,比如盐类物质和核苷酸等(详细纯化步骤及试剂盒原理请参照试剂盒说明书介绍),以便第二步PCR扩增获取更好的扩增效果。
第二轮PCR扩增以上述第一轮PCR产物经纯化稀释后的样品为模板注1,以341F/907R(341F 5’-CCTACGGGAGGCAGCAG-3’;907R(5’-CCGTCAATTCCTTTGAGTTT-3’)为扩增引物,进行第二轮扩增注2。25 μL反应为:Premix(5 U·μL-1,ExTaq,Takara)12.5 μL、341F/907R(20 pmol·μL-1)各1 μL、第一轮PCR产物1 μL、无菌水定容至25 μL 扩增条件为:94°C 3 min、94°C1 min、54°C1 min、72°C2 min(30个循环注3)、72°C10 min。
采用该方法所得的PCR产物可有效排除来自植物细胞器SSU rRNA基因的干扰,可进行高通量测序等下游实验,而且后续结果分析中也不需要进一步人工筛选不属于植物内生细菌的序列。
表1 LNA-PCR扩增引物和细菌特异性扩增引物
Table 1 Sequences of the designed LNA oligonucleotides and bacterial primers specific for the SSU rRNA genes from plant mitochondria and plastids
|
LNA扩增引物和细菌 特异性扩增引物 LNA oligonucleotide and bacterial primers |
序列 Sequence |
长度 Length (bases) |
参考文献 Reference |
|
|
LNA oligonucleotide |
LNA-Mit63 |
5’-GTCGAACGTTGTTTTCGGp-3’ |
18 |
(Ikenaga and Sakai 2014) |
|
LNA-Mit1492 |
5’-CTTCACCCCAGTCGAAGAp-3’ |
18 |
(Ikenaga and Sakai 2014) |
|
|
LNA-Pla63(大豆) |
5’-TCGGACGGGAAGTGGTp-3’ |
16 |
(Yu等, 2016) |
|
|
LNA-Pla1492(大豆) |
5’-CTTCACTCCAGTCACTAGCp-3’ |
19 |
(Yu等, 2016) |
|
|
LNA-Pla63(玉米) |
5’-GTCGAACGGGAAGTGGTp-3’ |
17 |
(Ikenaga and Sakai 2014) |
|
|
LNA-Pla1492(玉米) |
5’-CTTCACTCCAGTCGCAAGCp-3’ |
19 |
(Ikenaga and Sakai 2014) |
|
|
Bacterial primer |
KU63f |
5’-GCYTWAYACATGCAAGTC-3’ |
18 |
(Ikenaga等, 2018) |
|
KU1494r |
5’-GGYTACCTTGTTACGAC-3’ |
17 |
(Ikenaga等, 2018) |
|
注:“粗体碱基”表示LNA碱基;“下划线碱基”表示LNA引物和细菌引物交叉部分;“p”表示3'末端为磷酸化处理。其中
注意事项:
注释:
结果与分析
采用此方法得到的第一轮PCR产物经电泳检测后,在未采用LNA引物进行PCR扩增的样品中来自植物叶绿体和线粒体的条带占有优势,相反,在采用LNA引物进行PCR扩增的样品中,来自植物内生细菌的条带占有优势,如下图所示: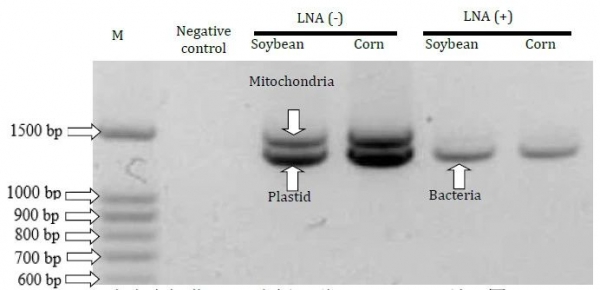
图2 以大豆和玉米内生细菌PCR为例,说明LNA-PCR效果图,marker(100 bp-1500 bp)Fig 2. Agarose gel electrophoresis ofthe PCR products amplified from the roots of soybean and corn with or withoutLNA oligonucleotides. M is the marker of 100 bp ladders.(图片来源:Yu et al.,2016)
以上述第一轮PCR产物为模板,采用带有barcode的细菌引物进行第二轮PCR扩增,再进行高通量测序后发现,对于大豆样品,采用LNA-PCR扩增后没有发现来自大豆叶绿体和线粒体的序列,而未采用LNA-PCR扩增后来自叶绿体和线粒体的序列分别占序列总数的66.43%和1.33%;对于玉米样品,采用LNA-PCR扩增后来自叶绿体和线粒体的序列分别占序列总数的0.13%和0.88%,而未采用LNA-PCR扩增后来自叶绿体和线粒体的序列分别占序列总数的93.15%和4.23%,进一步说明本方法在研究植物内生细菌多样性方面的优势。
致谢
1.感谢中国科学院战略先导对本工作的支持(XDB15010103);
2.感谢日本鹿儿岛大学Masao Sakai和Makoto Ikenaga对实验技术的指导;
3.已发表的使用过本实验方案的文章详见Ikenaga et al., 2014(Microbes and Environments, 29, 286–295);Ikenaga et al., 2015(Bioscience, Biotechnology, and Biochemistry, 79, 1556–1566);Yu et al., 2016(Journal of Integrative Agriculture 2016, 15(8): 1883–1891);Ikenaga et al., 2018(Microbes and Environments, 33, 340–344);喻江等(应用生态学报,2016,27(8) : 2663-2669);喻江(东北农业大学博士学位论文, 2016)。
参考文献
1.Ikenaga, M., Katsuragi, S., Handa, Y., Katsumata, H., Chishaki, N., Kawauchi, T. and Sakai, M. (2018). Improvements in Bacterial Primers to Enhance Selective SSU rRNA Gene Amplification of Plant-associated Bacteria by Applying the LNA Oligonucleotide-PCR Clamping Technique. Microbes and Environments 33(3): 340-344. https://doi.org/10.1264/jsme2.ME18071
2.Ikenaga, M. and Sakai, M. (2014). Application of Locked Nucleic Acid (LNA) Oligonucleotide-PCR Clamping Technique to Selectively PCR Amplify the SSU rRNA Genes of Bacteria in Investigating the Plant-Associated Community Structures. Microbes and Environments 29(3): 286-295. https://doi.org/10.1264/jsme2.ME14061
3.Yu, Z.-H., Yu, J., Ikenaga, M., Sakai, M., Liu, X.-B. and Wang, G.-H. (2016). Characterization of root-associated bacterial community structures in soybean and corn using locked nucleic acid (LNA) oligonucleotide-PCR clamping and 454 pyrosequencing. Journal of Integrative Agriculture 15(8): 1883-1891. https://doi.org/https://doi.org/10.1016/S2095-3119(15)61195-9