小麦(Triticum aestivum L.)是全球最重要的粮食作物之一。由于其基因组庞大、高度重复且为异源六倍体,导致其完整组装长期面临挑战。2018年,国际小麦基因组测序联盟(IWGSC)发布了中国春小麦参考基因组,成为世界范围内小麦研究应用最为广泛的参考基因组,为小麦基因组学研究的深入开展以及育种改良工作提供了关键支撑,极大地推动了相关领域的进步。然而,当下该基因组组装中仍存在大量未解析的重复区域和复杂序列结构,不仅使得小麦基因组学研究难以全方位覆盖整个基因组,还阻碍了针对这些特定区域开展精细化、深入的解析,制约了小麦基因组学研究的发展和育种应用。
2025年2月13日,傅向东研究组和鲁非研究组联合中国农业大学农学院在Molecular Plant在线发表了题为“Near-complete assembly and comprehensive annotation of the wheat Chinese Spring genome”的研究论文。本研究综合利用ONT超长读长测序(覆盖度283.56×)、PacBio HiFi高精度测序(29.01×)和Hi-C数据,实现了小麦中国春基因组的近完整组装(CS-CAU),其大小为14.46 Gb,碱基准确率大于99.9963%,仅剩290个组装间隙(主要为超长串联重复序列)。其中,1D、3D、4D、5D染色体首次实现无间隙组装,1D和5D染色体达到端粒到端粒(T2T)级别。这一突破不仅解决了小麦基因组重复序列高、多倍体复杂的组装难题,还为解析其他复杂作物基因组提供了范本。基于近完整基因组组装,研究团队总共注释到151,405个高置信度基因,其中59,180个是新注释的基因,包括7,602个首次组装出的基因,这对小麦基因功能研究具有重要意义。通过整合RNA-seq数据集和跨物种蛋白同源性证据,首次完整解析了六类种子储藏蛋白(SSP)的基因组分布与表达特征。研究发现,ω-醇溶蛋白的表达完全由B亚基因组贡献,而其他五类SSP(α/γ-醇溶蛋白、ALP、HMW/LMW谷蛋白)的表达则主要由D亚基因组贡献,为进一步解析小麦面筋品质的遗传基础和分子改良提供了重要基础。除chr1B的着丝粒存在与超长GAA重复序列相关的间隙外,其余20条染色体的着丝粒序列也都全部组装完成。对着丝粒区序列组成进行分析表明着丝粒区域主要由逆转座子构成,其中A/B亚基因组着丝粒富含着丝粒相关反转录转座子CRW和Quinta(占比约70%),而D亚基因组着丝粒中只有30%的序列为CRW和Quinta。相似的是,串联重复序列在三个亚基因组间分布也存在高度的不均匀性,其中71.89%的简单串联重复(SSR)富集于B亚基因组,而接近一半的卫星序列(satellite)则集中于D亚基因组。此外,研究团队也对着丝粒区CRW和Quinta逆转座子的插入时间进行了解析,明确了其在三个亚基因组间的主要扩张时期。
该研究利用牛津纳米孔(ONT)超长读长、PacBio HiFi高精度测序及Hi-C技术,成功构建了中国春小麦基因组的近完整组装(CS-CAU),几乎填补了此前基因组中所有的空白区域,为小麦遗传改良和基础研究提供了关键资源。中国农业大学农学院玉米改良中心陈建副教授、小麦研究中心孙其信院士、倪中福教授,中国科学院遗传与发育生物学研究所傅向东研究员、鲁非研究员为该论文的共同通讯作者。中国农业大学博士后王子健和博士研究生苗凌峰为论文共同第一作者。中国农业大学博士研究生谭凯文对该工作的推进有重要贡献。中国农业大学玉米改良中心赖锦盛教授、辛蓓蓓副教授,小麦研究中心郭伟龙教授,中国农业科学院作物科学研究所贾继增研究员,澳大利亚墨尔本大学Rudi Appels教授对该工作进行了指导和帮助。该工作得到了国家重点研发计划、国家自然科学基金项目、“拼多多-中国农业大学研究基金”、新基石研究员项目和中国农业大学2115人才培育发展支持计划的资助。
文章链接:https://doi.org/10.1016/j.molp.2025.02.002
CS-CAU基因组下载路径:https://www.ncbi.nlm.nih.gov/nuccore/JBJQUP000000000.1 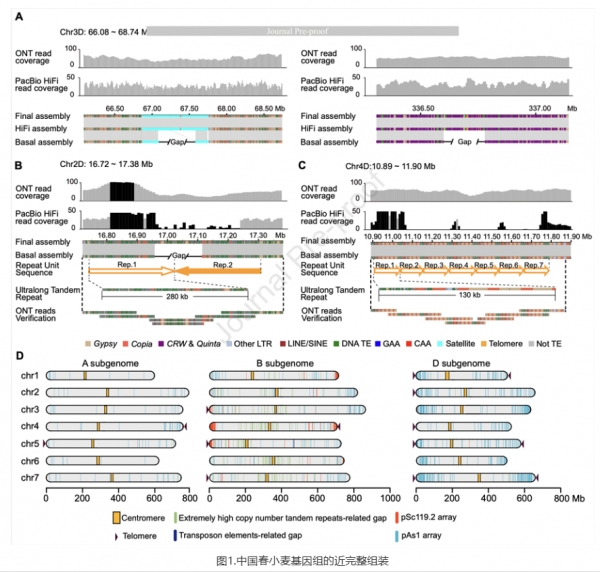
图1.中国春小麦基因组的近完整组装