普通小麦是异源六倍体,其基因组规模庞大且结构复杂,包含A、B、D三套高度同源的染色体组,这使得在其中精确定位特定基因变得极为困难。传统的遗传定位方法,如基于自然群体的GWAS和基于双亲杂交群体QTL定位等,在小麦中的应用效率和精度远不如在二倍体作物(如水稻)中理想,主要受限于小麦同源序列的功能冗余。同时,在小麦的长期驯化和育种选择过程中,差异性位点(如SNP、InDel等)被偏好性选择,并以单倍型形式存在于自然群体中,这进一步限制了基因的正向克隆和功能研究。尽管EMS(甲基磺酸乙酯)诱变产生的近饱和突变体库因其突变随机、突变位点覆盖度高、能打破群体结构限制并克服连锁不平衡的影响等优势被广泛用于不同物种的功能基因克隆和机制研究中,但目前基于EMS突变体库开展的高通量、规模化的小麦功能基因定位研究尚未取得实质性进展。此外,传统小麦农艺性状(如千粒重、株高、分蘖数等)的调查通常费时费力,而一些难以通过人工观测获取的新颖几何性状(如小穗角度、小穗间距),同样是影响小麦产量的关键因素。对这些几何性状进行解析与功能研究,将为挖掘小麦抗逆与高产的潜在基因资源提供新的途径。
近日,中国科学院遗传与发育生物学研究所育种前沿技术实验室贺飞研究组、蒋霓研究组联合河北师范大学生命科学学院的小麦研究团队突破了利用小麦EMS群体高通量挖掘功能基因的技术瓶颈,结合高通量表型组学技术开发了适用于EMS突变体库的全基因组关联分析的新方法GH-GLA(GeneHunter-Gene-Level Association)。该方法实现了高效鉴定小麦重要农艺性状相关的候选基因,为挖掘小麦功能基因和解析基因作用机制提供大量资源和线索。相关研究A forward genetics strategy for high-throughput gene identification via precise image-based phenotyping of an indexed EMS mutant library于2025年9月29日在线发表于Advanced Science杂志。
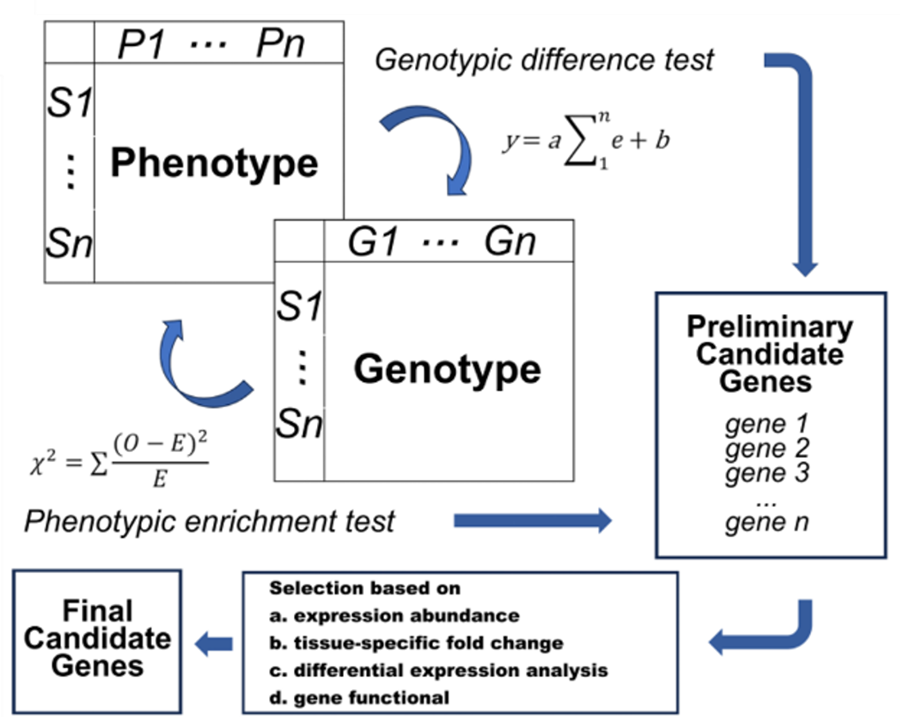
本研究以河北师范大学李俊明教授(原中国科学院遗传与发育生物学研究所研究员)培育的“科农9204”为材料,在前期构建的EMS近饱和突变体库基础上(Wang et al., 2023, Plant Communications),利用深度学习驱动的图像表型鉴定平台(Sun et al., 2025, Plant Phenomics),对近2000个突变株系进行了高通量图像采集和表型分析,共提取了83个株型和穗部性状。为解决EMS突变体中低频突变的分析难题,团队基于该突变体库的外显子捕获测序数据,优化了分析策略并开发了GH-GLA流程。该流程将分析单元从“单个突变位点”提升至“整个基因”,从而显著提高了突变体库的利用效率。
通过GH-GLA方法,研究团队从小麦中鉴定出5905个与重要性状相关的候选基因,并利用CRISPR/Cas9技术成功验证了其中3个基因的功能:TaAN-1(调控千粒重)、TaBAM5L(影响粒型与千粒重)和TaXTH28L(调控穗型)。此外,基于小麦微核心种质自然群体的遗传分析进一步揭示了以下单倍型与千粒重和高产的相关性:TaAN-1-2B的Hap3、TaBAM5L-5A的Hap2以及TaXTH28L-4A的Hap3。这些单倍型具有显著的育种潜力,从而证明了GH-GLA方法在候选基因功能预测上的有效性。
GH-GLA方法分析流程